
Notre recherche
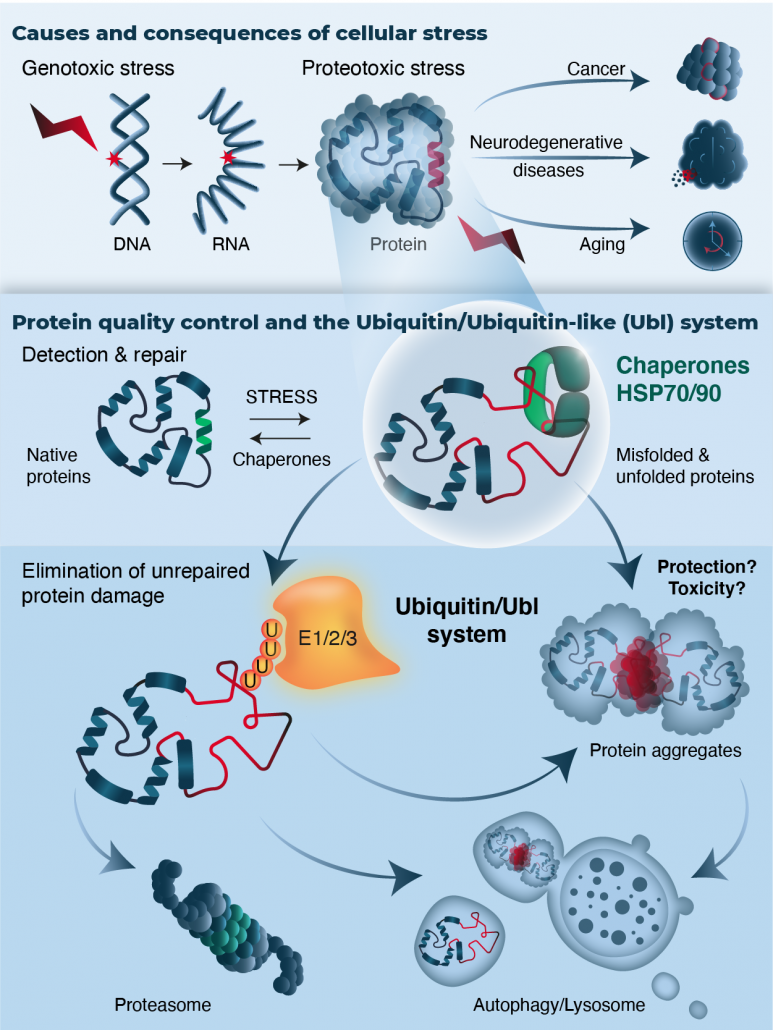
Le système de contrôle de la qualité des protéines (PQC)
Comprendre comment les organismes réagissent au stress environnemental a des implications cruciales sur la qualité de vie et le traitement des maladies. Le maintien d’un protéome fonctionnel est un défi pour les cellules étant donné la variété des stress protéotoxiques (exposition aux produits chimiques, température élevée, espèces oxydantes) qui causent constamment des dommages aux protéines.
Les protéines mal repliées sont délétères pour la cellule en raison de la perte de leur fonction et/ou de leur capacité à former des agrégats insolubles. La génération d’inclusion protéiques aberrants (agrégat) est observée au cours du vieillissement et a été associée à de multiples pathologies connues sous le nom de « désordres conformationnels ». Il s’agit notamment des maladies lysosomales, de la mucoviscidose, de certain cancer et de nombreuses maladies neurodégénératives telles que les maladies d’Alzheimer, de Huntington, de Parkinson et la Sclérose Latérale Amyotrophique (SLA).
Pour contrer et prévenir le mauvais repliement et l’agrégation des protéines, les cellules ont développé un réseau de contrôle de la qualité des protéines (PQC). Des molécules chaperonnes coopèrent avec des mécanismes protéolytiques (protéasome 26S, voie lysosomale) pour réparer, replier ou éliminer des protéines endommagées, maintenant ainsi la protéostasie.
La gestion spatiale des protéines mal repliées dans le cytoplasme et le noyau semble être crucial au fonctionnement du système PQC. En effet, la séquestration des protéines mal repliées dans des compartiments subcellulaires particuliers facilite l’élimination des protéines endommagées. Un équilibre doit être respecté pour avoir une élimination efficace tout en évitant la génération d’agrégats protéiques.
L’Ubiquitine et les molécules apparentées à l’Ubiquitine, régulateurs clés du système PQC
La famille de l’Ubiquitine et des molécules apparentées à l’Ubiquitine (Ubls), telles que SUMO et NEDD8, joue un rôle essentiel dans tous les aspects du système PQC. L’élimination de la plupart des protéines aberrantes est assurée par le système Ubiquitine/Protéasome (UPS) : Un mécanisme complexe permet de fixer de façon covalente des molécules d’Ubiquitine aux protéines endommagées, qui seront alors dégradées dans le protéasome. La famille des Ubls apparaît comme un régulateur important de la signalisation de l’Ubiquitine et du système PQC.
Nos études ont révélé des rôles distincts pour la molécule Ubiquitin-like NEDD8 lors de stress protéotoxiques. Nous avons mis en avant que la formation de chaînes poly-NEDD8 dans le cytoplasme et de chaînes hybrides NEDD8-Ubiquitine dans le noyaux contrôlait la formation d’inclusions protéiques. Notre objectif est de décrypter le rôle de NEDD8 lors de stress sur les protéines mal repliées et la formation d’agrégats protéiques. Nous combinons des approches biochimiques, de biologie cellulaire, d’imagerie et de protéomique sur des cellules humaines et C.elegans pour étudier au mieux ces mécanismes sous-jacents. La voie NEDD8 semble être directement impliqué dans de nombreuse pathologie, ce qui fait d’elle une cible thérapeutique prometteuse. La compréhension des mécanismes de réponse au stress pourrait permettre d’identifier de nouvelles cibles au sein du système PQC, en vue d’une intervention thérapeutique ciblé grâce à des inhibiteurs de la voie NEDD8 spécifique.
En savoir plus
Financements
ANR PROCONUC (Dimitris Xirodimas)
Publications
2024
- A nuclear protein quality control system for elimination of nucleolus-related inclusions. Brunello L, Polanowska J, Le Tareau L, Maghames C, Georget V, Guette C, Chaoui K, Balor S, O’Donohue MF, Bousquet MP, Gleizes PE, Xirodimas DP. EMBO J. 2024 Dec 17, Epub ahead of print Pubmed
- An Anti-Invasive Role for Mdmx through the RhoA GTPase under the Control of the NEDD8 Pathway. Bou Malhab LJ, Schmidt S, Fagotto-Kaufmann C, Pion E, Gadea G, Roux P, Fagotto F, Debant A, Xirodimas DP. Cells. 2024 Sep 28;13(19):1625 Pubmed
- Neddylation inhibition prevents acetaminophen-induced liver damage by enhancing the anabolic cardiolipin pathway.. Gil-Pitarch C, Serrano-Maciá M, Simon J, Mosca L, Conter C, Rejano-Gordillo CM, Zapata-Pavas LE, Peña-Sanfélix P, Azkargorta M, Rodríguez-Agudo R, Lachiondo-Ortega S, Mercado-Gómez M, Delgado TC, Porcelli M, Aurrekoetxea I, Sutherland JD, Barrio R, Xirodimas D, Aspichueta P, Elortza F, Martínez-Cruz LA, Nogueiras R, Iruzubieta P, Crespo J, Masson S, McCain MV, Reeves HL, Andrade RJ, Lucena MI, Mayor U, Goikoetxea-Usandizaga N, González-Recio I, Martínez-Chantar ML. Cell Rep Med. 2024 Jul 16;5(7):101653. Pubmed
- SUMOylation modulates eIF5A activities in both yeast and pancreatic ductal adenocarcinoma cells. Seoane R, Lama-Díaz T, Romero AM, El Motiam A, Martínez-Férriz A, Vidal S, Bouzaher YH, Blanquer M, Tolosa RM, Castillo Mewa J, Rodríguez MS, García-Sastre A, Xirodimas D, Sutherland JD, Barrio R, Alepuz P, Blanco MG, Farràs R, Rivas C. Cell Mol Biol Lett. 2024 Jan 16;29(1):15. Pubmed
2023
- Two distinct mechanisms lead to either oocyte or spermatocyte decrease in C. elegans after whole developmental exposure to γ-rays. Dufourcq Sekatcheff E, Godon C, Bailly A, Quevarec L, Camilleri V, Galas S, Frelon S. PLoS One. 2023 Nov 27;18(11):e0294766. Pubmed
- Deneddylation of ribosomal proteins promotes synergy between MLN4924 and chemotherapy to elicit complete therapeutic responses. Aubry A, Pearson JD, Charish J, Yu T, Sivak JM, Xirodimas DP, Avet-Loiseau H, Corre J, Monnier PP, Bremner R. Cell Rep. 2023 Aug 7;42(8):112925. Pubmed
- Targeting the NEDP1 enzyme to ameliorate ALS phenotypes through stress granule disassembly. Kassouf T, Shrivastava R, Meszka I, Bailly A, Polanowska J, Trauchessec H, Mandrioli J, Carra S, Xirodimas DP. Sci Adv. 2023 Mar 31;9(13):eabq7585. Pubmed
- A Mass Spectrometry-Based Strategy for Mapping Modification Sites for the Ubiquitin-Like Modifier NEDD8. Oliveira CAB, Isaakova E, Beli P, Xirodimas DP. Methods Mol Biol. 2023;2602:137-149. Pubmed
2022
- Mixed in chains: NEDD8 polymers in the Protein Quality Control system. Meszka I, Polanowska J, Xirodimas DP. Semin Cell Dev Biol. 2022 Dec;132:27-37. Pubmed
2021
- Neddylation inhibition ameliorates steatosis in NAFLD by boosting hepatic fatty acid oxidation via the DEPTOR-mTOR axis. Serrano-Maciá M, Simón J, González-Rellan MJ, Azkargorta M, Goikoetxea-Usandizaga N, Lopitz-Otsoa F, De Urturi DS, Rodríguez-Agudo R, Lachiondo-Ortega S, Mercado-Gomez M, Gutiérrez de Juan V, Bizkarguenaga M, Fernández-Ramos D, Buque X, Baselli GA, Valenti LVC, Iruzubieta P, Crespo J, Villa E, Banales JM, Avila MA, Marin JJG, Aspichueta P, Sutherland J, Barrio R, Mayor U, Elortza F, Xirodimas DP, Nogueiras R, Delgado TC, Martínez-Chantar ML. Mol Metab. 101275 Pubmed
- The HSP70 chaperone as sensor of the NEDD8 cycle upon DNA damage. Bailly AP, Xirodimas DP. Biochem Soc Trans. 49(3):1075-1083. Pubmed
- Proteome-wide identification of NEDD8 modification sites reveals distinct proteomes for canonical and atypical NEDDylation. Sofia Lobato-Gil, Jan B. Heidelberger, Chantal Maghames, Aymeric Bailly, Lorene Brunello, Manuel S. Rodriguez, Petra Beli, and Dimitris P. Xirodimas. Cell Rep. 34(3):108635. Pubmed
2020
- Development of Nanobodies as first-in-class inhibitors for the NEDP1 deNEDDylating enzyme. Naima Abidi, Helene Trauchessec, Gholamreza Hassanzadeh-Ghassabeh, Martine Pugniere, Serge Muyldermans, Dimitris P. Xirodimas. bioRxiv 2020.03.20.999326 Pubmed
- Antibody RING-Mediated Destruction of Endogenous Proteins. Ibrahim AFM, Shen L, Tatham MH, Dickerson D, Prescott AR, Abidi N, Xirodimas DP, Hay RT. Mol Cell ; S1097-2765(20)30274-4. Pubmed
2019
- The balance between Mono- and NEDD8-Chains Controlled by NEDP1 upon DNA Damage Is a Regulatory Module of the HSP70 ATPase Activity. Bailly AP, Perrin A, Serrano-Macia M, Maghames C, Leidecker O, Trauchessec H, Martinez-Chantar ML, Gartner A, Xirodimas DP. Cell Rep. 29(1):212-224 Pubmed
2018
- NEDDylation promotes nuclear protein aggregation and protects the Ubiquitin Proteasome System upon proteotoxic stress. Maghames CM, Lobato-Gil S, Perrin A, Trauchessec H, Rodriguez MS, Urbach S, Marin P, Xirodimas DP. Nat Commun. 9(1):4376. Pubmed
- Interplay between SUMOylation and NEDDylation regulates RPL11 localization and function. El Motiam A, Vidal S, de la Cruz-Herrera CF, Da Silva-Álvarez S, Baz-Martínez M, Seoane R, Vidal A, Rodríguez MS, Xirodimas DP, Carvalho AS, Beck HC, Matthiesen R, Collado M, Rivas C. FASEB J. :fj201800341RR Pubmed
2017
- Quantitative FLIM-FRET Microscopy to Monitor Nanoscale Chromatin Compaction In Vivo Reveals Structural Roles of Condensin Complexes. Llères D, Bailly AP, Perrin A, Norman DG, Xirodimas DP, Feil R. Cell Rep. 18:1791-1803. Pubmed
2016
- The use of the NEDD8 inhibitor MLN4924 (Pevonedistat) in a cyclotherapy approach to protect wild-type p53 cells from MLN4924 induced toxicity. Malhab LJ, Descamps S, Delaval B, Xirodimas DP. Sci Rep. 6:37775. Pubmed
- The NEDD8 inhibitor MLN4924 increases the size of the nucleolus and activates p53 through the ribosomal-Mdm2 pathway. Bailly A, Perrin A, Bou Malhab LJ, Pion E, Larance M, Nagala M, Smith P, O’Donohue MF, Gleizes PE, Zomerdijk J, Lamond AI, Xirodimas DP. 35:415-26. Pubmed
2015
- PKA-dependent phosphorylation of ribosomal protein S6 does not correlate with translation efficiency in striatonigral and striatopallidal medium-sized spiny neurons. Biever A, Puighermanal E, Nishi A, David A, Panciatici C, Longueville S, Xirodimas D, Gangarossa G, Meyuhas O, Hervé D, Girault JA, Valjent E. J Neurosci. 35:4113-30. Pubmed
- Regulation of cancer-related pathways by protein NEDDylation and strategies for the use of NEDD8 inhibitors in the clinic. Abidi N, Xirodimas DP. Endocr Relat Cancer. 22:T55-70. Pubmed
2013
- DNA-binding regulates site-specific ubiquitination of IRF-1. Landré V, Pion E, Narayan V, Xirodimas DP, Ball KL. Biochem J. 449(3):707-17. Pubmed
- DUBs « found in translation »: USP15 controls stability of newly synthesized REST. Xirodimas DP. Cell Cycle. 12(16):2536-7. Pubmed
2012
- Characterization of MRFAP1 turnover and interactions downstream of the NEDD8 pathway. Larance M, Kirkwood KJ, Xirodimas DP, Lundberg E, Uhlen M, Lamond AI. Mol Cell Proteomics. 11(3):M111.014407. Pubmed
- Murine double minute 2 regulates Hu antigen R stability in human liver and colon cancer through NEDDylation. Embade N, Fernández-Ramos D, Varela-Rey M, Beraza N, Sini M, Gutiérrez de Juan V, Woodhoo A, Martínez-López N, Rodríguez-Iruretagoyena B, Bustamante FJ, de la Hoz AB, Carracedo A, Xirodimas DP, Rodríguez MS, Lu SC, Mato JM, Martínez-Chantar ML. 55(4):1237-48. Pubmed
- Isolation of NEDDylated proteins in human cells. Leidecker O, Xirodimas DP. Methods Mol Biol. 832:133-40. Pubmed
- The ubiquitin E1 enzyme Ube1 mediates NEDD8 activation under diverse stress conditions. Leidecker O, Matic I, Mahata B, Pion E, Xirodimas DP. Cell Cycle. 11(6):1142-50. Pubmed
- The p53 isoforms are differentially modified by Mdm2. Camus S, Ménendez S, Fernandes K, Kua N, Liu G, Xirodimas DP, Lane DP, Bourdon JC. Cell Cycle.11(8):1646-55. Pubmed
- Influence of the nuclear membrane, active transport, and cell shape on the Hes1 and p53-Mdm2 pathways: insights from spatio-temporal modelling. Sturrock M, Terry AJ, Xirodimas DP, Thompson AM, Chaplain MA. Bull Math Biol. 74(7):1531-79. Pubmed
2011
- Spatio-temporal modelling of the Hes1 and p53-Mdm2 intracellular signalling pathways. Sturrock M, Terry AJ, Xirodimas DP, Thompson AM, Chaplain MA. J Theor Biol. 273(1):15-31. Pubmed
- The essential functions of NEDD8 are mediated via distinct surface regions, and not by polyneddylation in Schizosaccharomyces pombe. Girdwood D, Xirodimas DP, Gordon C. PLoS One. 6(5):e20089. Pubmed
- In the family with ubiquitin. Alexandru G, Pariente N, Xirodimas D. EMBO Rep. 12(9):880-2. Pubmed
- Stable-isotope labeling with amino acids in nematodes. Larance M, Bailly AP, Pourkarimi E, Hay RT, Buchanan G, Coulthurst S, Xirodimas DP, Gartner A, Lamond AI. Nat Methods. 8(10):849-51. Pubmed
2010
- Perturbation of 60 S ribosomal biogenesis results in ribosomal protein L5- and L11-dependent p53 activation. Sun XX, Wang YG, Xirodimas DP, Dai MS. J Biol Chem. 285(33):25812-21. Pubmed
- Mechanism of hypoxia-induced NF-kappaB. Culver C, Sundqvist A, Mudie S, Melvin A, Xirodimas D, Rocha S. Mol Cell Biol. 30(20):4901-21. Pubmed
- Ubiquitin Family Members in the Regulation of the Tumor Suppressor p53. Xirodimas DP, Scheffner M. Subcell Biochem. 54:116-35. Pubmed
2009
- Regulation of nucleolar signalling to p53 through NEDDylation of L11. Sundqvist A, Liu G, Mirsaliotis A, Xirodimas DP. EMBO Rep. 10(10):1132-9. Pubmed
- Detection of protein SUMOylation in vivo. Tatham MH, Rodriguez MS, Xirodimas DP, Hay RT. Nat Protoc. 4(9):1363-71. Pubmed
2008
- Ribosomal proteins are targets for the NEDD8 pathway. Xirodimas DP, Sundqvist A, Nakamura A, Shen L, Botting C, Hay RT. EMBO Rep. 9(3):280-6. Pubmed
- Much to know about proteolysis: intricate proteolytic machineries compromise essential cellular functions. Marfany G, Farràs R, Salido E, Xirodimas DP, Rodríguez MS. Biochem Soc Trans. 36(Pt 5):781-5. Pubmed
- Novel substrates and functions for the ubiquitin-like molecule NEDD8. Xirodimas DP. Biochem Soc Trans. 36(Pt 5):802-6. Pubmed
Ubiquitine / Molécules de type Ubiquitine et contrôle de la qualité des protéines
 Dimitris XIRODIMAS
Dimitris XIRODIMAS
Chef d’équipe (Chercheur DR2 CNRS)
Membres de l’équipe
(AI-Recherche) +33 (0)4 34 35 95 33 |
|
(Chercheur DR2) +33 (0)4 34 35 95 33 |
|
(CRCN) +33 (0)4 34 35 95 34 |
|
(CRCN) +33 (0)4 34 35 95 33 |
|
(Chercheur) +33 (0)4 34 35 95 34 |
|
(Doctorant) +33 (0)4 34 35 95 33 |
|
(Doctorant) +33 (0)4 34 35 95 33 |
|
(Stagiaire) +33 (0)4 34 35 95 34 |

Contactez notre équipe
Remplacez le nom et l’adresse ci-dessus par celui du membre à contacter
prenom.nom@crbm.cnrs.fr






