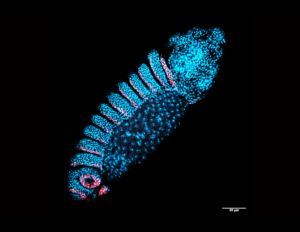
Notre Recherche
Le développement de l’embryon est un processus de non-équilibre présentant des comportements émergents : les interactions moléculaires à l’intérieur des cellules façonnent de manière irréversible les changements morphologiques du corps de l’animal en régulant les gènes du développement. Pour initier et contrôler l’expression des gènes, les facteurs de transcription (TF) doivent se lier aux enhancers et aux promoteurs. Cependant, des interactions transitoires et de faible affinité entre les TF et les enhancers du développement semblent être répandues et même nécessaires à la spécificité de la régulation chez les animaux, des nématodes aux mammifères. Comment ces faibles interactions pourraient-elles produire de manière fiable les schémas d’expression précis qui sont nécessaires pour contrôler l’embryogenèse ?
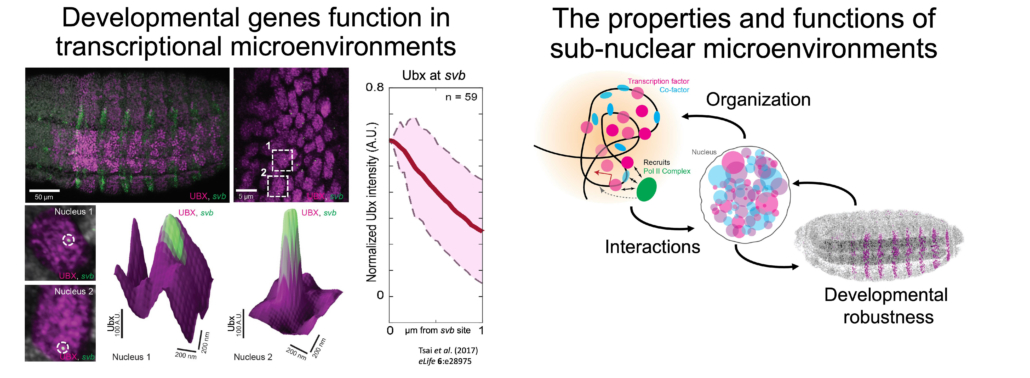
En utilisant des embryons de Drosophila melanogaster, nos travaux ont montré que des TFs tels que le facteur Homeobox (Hox) Ultrabithorax (Ubx) se regroupent en microenvironnements sub-nucléaires spécialisés autour du gène de développement shavenbaby (svb). Plusieurs gènes peuvent partager et renforcer le même environnement, améliorant ainsi la robustesse de l’expression génétique et du développement du phénotype face aux perturbations environnementales et génétiques. Ainsi, l’organisation spatiale des TFs et des gènes à l’intérieur du noyau pourrait être une stratégie pour faciliter la robustesse du développement. Cependant, on sait peu de choses sur les propriétés physiques et interactionnelles de ces environnements. Quand se forment-ils au cours de l’embryogenèse ? Qu’est-ce qui détermine leur emplacement et leur composition ? Quels rôles joue la chromatine ? Comment les gènes interagissent-ils avec les micro-environnements ? Comment affectent-ils les comportements des TFs ?
Pour répondre à ces questions, notre groupe combine les outils génétiques disponibles chez Drosophila melanogaster avec l’imagerie de fluorescence à haute et super-résolution pour étudier l’interaction des gènes et des TFs dans les microenvironnements transcriptionnels dans le contexte du développement embryonnaire.
1) Quand les micro-environnements se forment-ils et comment l’organisation nucléaire guide-t-elle la différenciation cellulaire ?
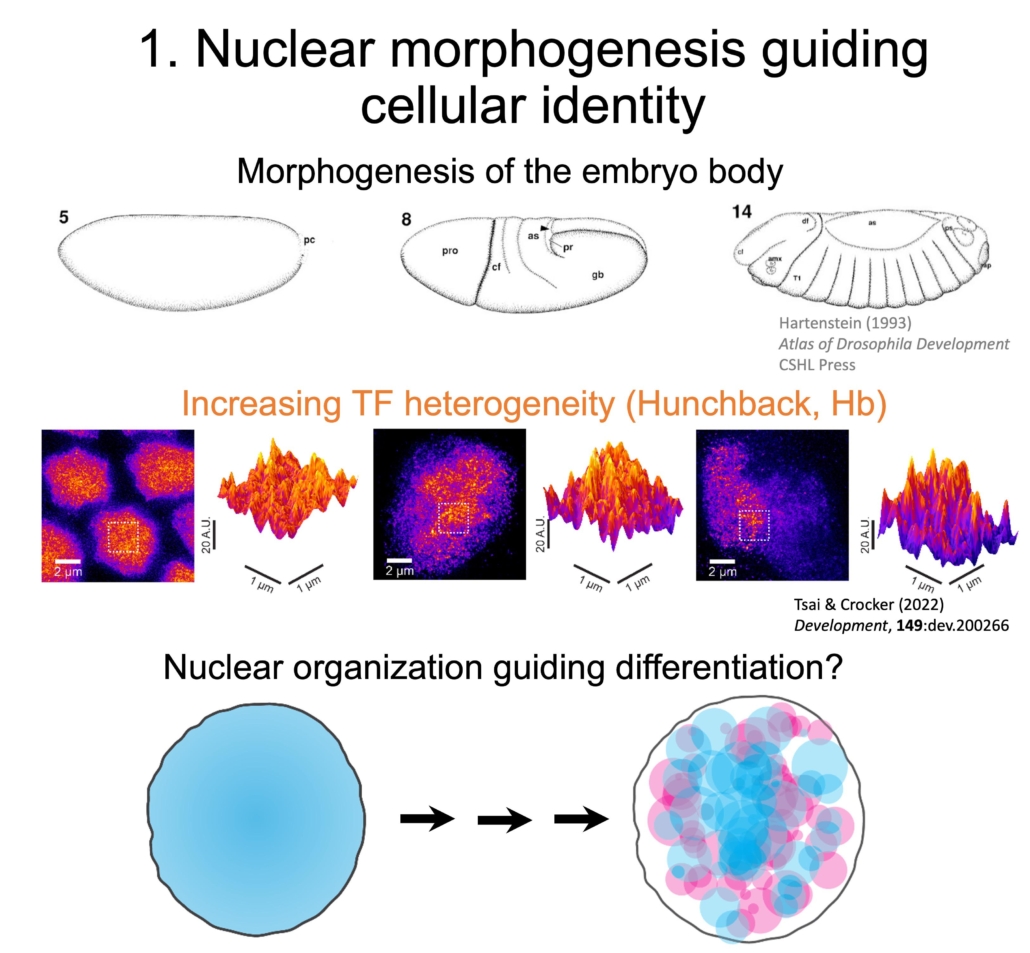
Au cours de la maturation de l’embryon, son corps subit une morphogenèse qui transforme l’œuf fécondé en un corps doté de structures complexes. Nous avons observé que les distributions nucléaires de plusieurs TFs deviennent de plus en plus hétérogènes au cours de cette même période. Les modifications des histones ont montré une augmentation similaire de l’hétérogénéité à mesure que le développement progresse. Cela suggère que l’espace nucléaire subit également une « morphogenèse » qui le partitionne en compartiments distincts.
Nous observons maintenant différentes lignées cellulaires au cours du développement afin de comparer comment les distributions des TF et des marques épigénétiques dans le noyau peuvent guider la différenciation.
2) La colocalisation peut-elle coordonner la corégulation des gènes dans les réseaux transcriptionnels ?
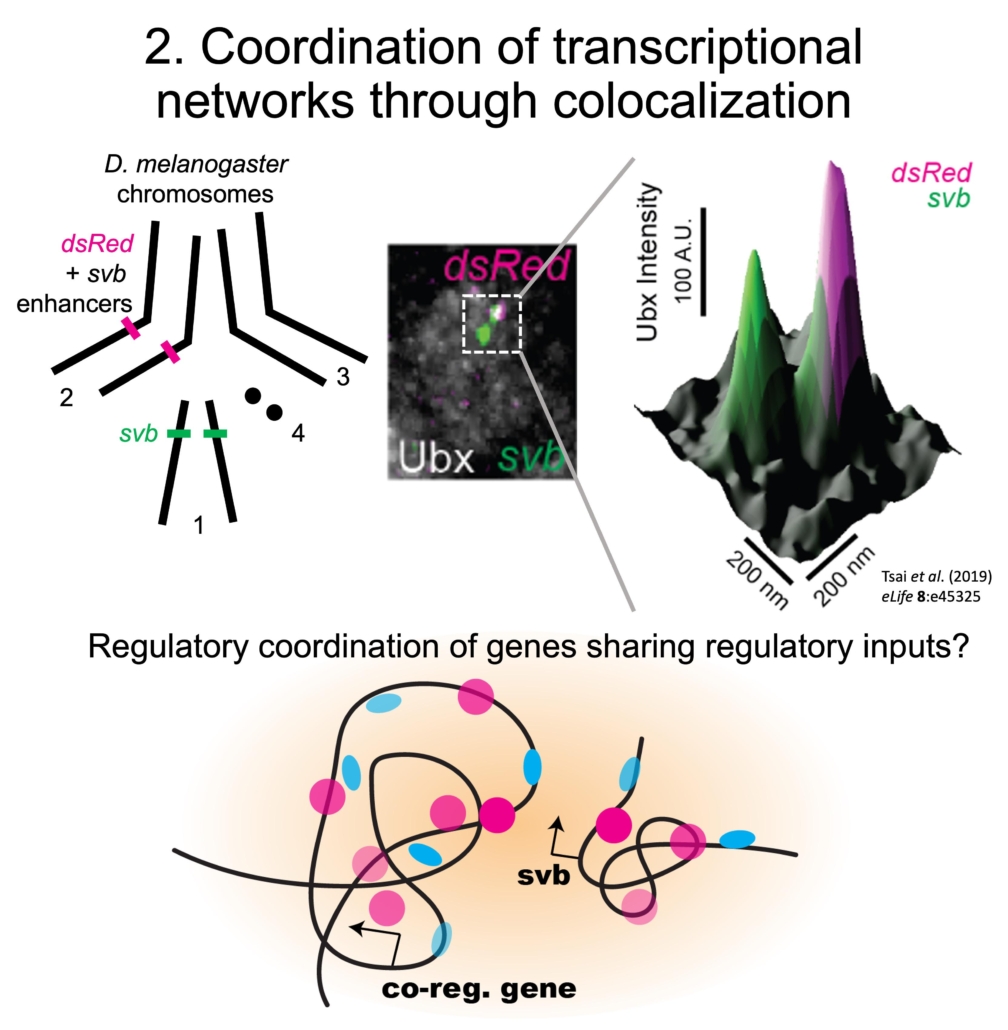
Les sites de transcription transgéniques dirigés par les exhausteurs svb se sont localisés avec le locus svb endogène et ont augmenté la concentration d’Ubx et la production transcriptionnelle de svb, ce qui a permis de sauver le phénotype du trichome dans une lignée de mouches avec un locus svb déficient. Ainsi, les gènes peuvent utiliser la proximité physique dans les micro-environnements pour améliorer leur coordination réglementaire et leur robustesse. Cependant, il n’est pas clair si la proximité physique des gènes et l’activité transcriptionnelle sont liées et si la colocalisation se produit entre d’autres gènes dans les réseaux transcriptionnels.
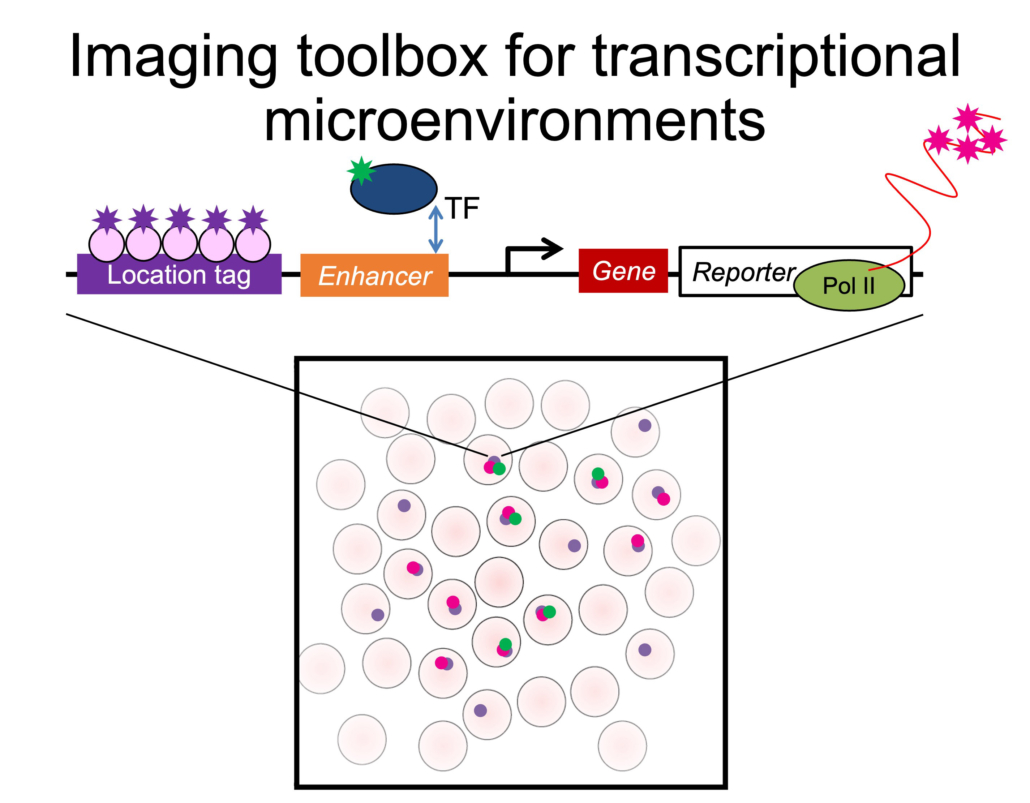
Ainsi, nous développons des outils d’imagerie dans les embryons pour surveiller la position et l’état transcriptionnel des gènes afin de comprendre la coordination spatiale des gènes co-régulés dans les réseaux de régulation.
3) Comment les interactions locales avec les protéines et l’ADN modifient-elles le comportement des TF ?
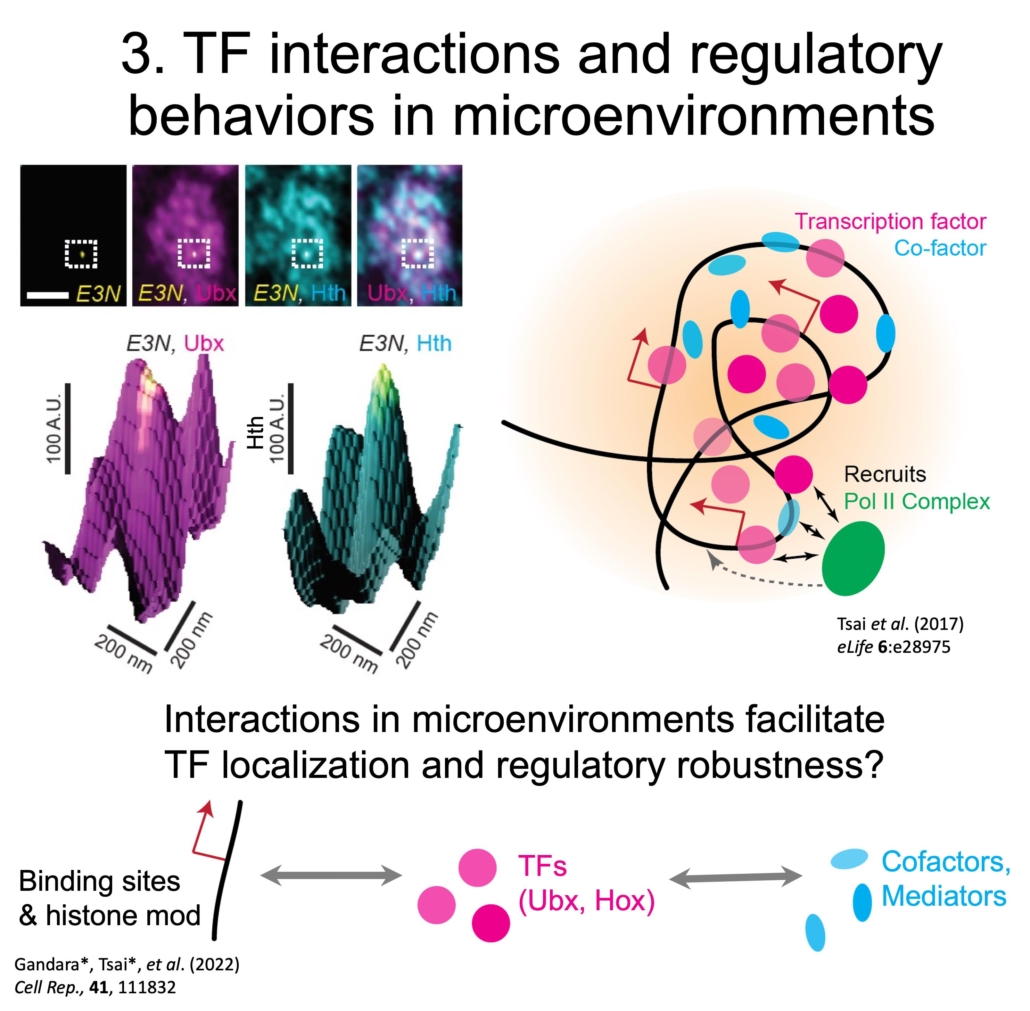
En général, les TF ne se colocalisent pas entre elles ni avec les marqueurs de l’activité transcriptionnelle, comme la PolII active (S5P). Cependant, les sites de transcription de svb ont eu tendance à être enrichis à la fois pour Ubx et pour un cofacteur spécifique, Homothorax (Hth). Cette différence entre le recouvrement global et local de différents facteurs suggère que les microenvironnements transcriptionnels sont formés en utilisant des interactions spécifiques. Il est intéressant de noter que Ubx et son paralogue de la famille Hox, AbdominalA (AbdA), ne se sont pas colocalisés, bien qu’ils aient des séquences consensus de liaison à l’ADN identiques. Cela suggère que des interactions autres que la liaison à l’ADN déterminent également la localisation des TFs.
Nous marquons donc les TFs du développement pour des expériences d’imagerie en direct et modifions leurs domaines d’interaction pour comprendre comment les TFs se localisent et comment leurs comportements régulateurs changent en fonction des interactions locales TF-ADN et TF-protéines dans les micro-environnements.
En savoir plus
Publications
2022
- Developmental phenomics suggests that H3K4 monomethylation confers multi-level phenotypic robustness. Gandara L, Tsai A, Ekelöf M, Galupa R, Preger-Ben Noon E, Alexandrov T, Crocker J. Cell Rep. 2022 Dec 13;41(11):111832. Pubmed
- Nuclear morphogenesis: forming a heterogeneous nucleus during embryogenesis. Tsai A, Crocker J. Development. 2022 Feb 15;149(4):dev200266. Pubmed
2020
- Dense and pleiotropic regulatory information in a developmental enhancer. Fuqua T, Jordan J, van Breugel ME, Halavatyi A, Tischer C, Polidoro P, Abe N, Tsai A, Mann RS, Stern DL, Crocker J. Nature. 2020 Nov;587(7833):235-239. Pubmed
- Robust and efficient gene regulation through localized nuclear microenvironments. Tsai A, Galupa R, Crocker J. Development. 2020 Oct 5;147(19):dev161430. Pubmed
2019
- Multi-enhancer transcriptional hubs confer phenotypic robustness. Tsai A, Alves MR, Crocker J. Elife. 2019 Jul 11;8:e45325. Pubmed
2018
- Visualizing long-range enhancer-promoter interaction. Tsai A, Crocker J. Nat Genet. 2018 Sep;50(9):1205-1206. Pubmed
- Transvection Goes Live-Visualizing Enhancer-Promoter Communication between Chromosomes. Tsai A, Singer RH, Crocker J. Mol Cell. 2018 Apr 19;70(2):195-196. Pubmed
2017
- Nuclear microenvironments modulate transcription from low-affinity enhancers. Tsai A, Muthusamy AK, Alves MR, Lavis LD, Singer RH, Stern DL, Crocker J. Elife. 2017 Nov 2;6:e28975. Pubmed
- A Fully Synthetic Transcriptional Platform for a Multicellular Eukaryote. Crocker J, Tsai A, Stern DL. Cell Rep. 2017 Jan 3;18(1):287-296. Pubmed
2016
- Rapid dynamics of general transcription factor TFIIB binding during preinitiation complex assembly revealed by single-molecule analysis. Zhang Z, English BP, Grimm JB, Kazane SA, Hu W, Tsai A, Inouye C, You C, Piehler J, Schultz PG, Lavis LD, Revyakin A, Tjian R. Genes Dev. 2016 Sep 15;30(18):2106-2118. Pubmed
- Glutamate-induced RNA localization and translation in neurons. Yoon YJ, Wu B, Buxbaum AR, Das S, Tsai A, English BP, Grimm JB, Lavis LD, Singer RH. Proc Natl Acad Sci U S A. 2016 Nov 1;113(44):E6877-E6886. 35:415-26. Pubmed`
- Amino acid sequence repertoire of the bacterial proteome and the occurrence of untranslatable sequences. Navon SP, Kornberg G, Chen J, Schwartzman T, Tsai A, Puglisi EV, Puglisi JD, Adir N. Proc Natl Acad Sci U S A. 2016 Jun 28;113(26):7166-70. Pubmed
2015
- Probing the Translation Dynamics of Ribosomes Using Zero-Mode Waveguides. Tsai A, Puglisi JD, Uemura S. Prog Mol Biol Transl Sci. 2016;139:1-43. Pubmed
2014
- Dynamic pathways of -1 translational frameshifting. Chen J, Petrov A, Johansson M, Tsai A, O’Leary SE, Puglisi JD. Nature. 2014 Aug 21;512(7514):328-32. Pubmed
- Sequence-dependent elongation dynamics on macrolide-bound ribosomes. Johansson M, Chen J, Tsai A, Kornberg G, Puglisi JD. Cell Rep. 2014 Jun 12;7(5):1534-1546. Pubmed
- The dynamics of SecM-induced translational stalling. Tsai A, Kornberg G, Johansson M, Chen J, Puglisi JD. Cell Rep. 2014 Jun 12;7(5):1521-1533. Pubmed
- Signal recognition particle-ribosome binding is sensitive to nascent chain length. Noriega TR, Tsai A, Elvekrog MM, Petrov A, Neher SB, Chen J, Bradshaw N, Puglisi JD, Walter P. J Biol Chem. 2014 Jul 11;289(28):19294-305. Pubmed
- High-throughput platform for real-time monitoring of biological processes by multicolor single-molecule fluorescence. Chen J, Dalal RV, Petrov AN, Tsai A, O’Leary SE, Chapin K, Cheng J, Ewan M, Hsiung PL, Lundquist P, Turner SW, Hsu DR, Puglisi JD. Proc Natl Acad Sci U S A. 2014 Jan 14;111(2):664-9. Pubmed
2013
- Involvement of protein IF2 N domain in ribosomal subunit joining revealed from architecture and function of the full-length initiation factor. Simonetti A, Marzi S, Billas IM, Tsai A, Fabbretti A, Myasnikov AG, Roblin P, Vaiana AC, Hazemann I, Eiler D, Steitz TA, Puglisi JD, Gualerzi CO, Klaholz BP. Proc Natl Acad Sci U S A. 2013 Sep 24;110(39):15656-61. Pubmed
- Coordinated conformational and compositional dynamics drive ribosome translocation. Chen J, Petrov A, Tsai A, O’Leary SE, Puglisi JD. Nat Struct Mol Biol. 2013 Jun;20(6):718-27. Pubmed
- The impact of aminoglycosides on the dynamics of translation elongation. Tsai A, Uemura S, Johansson M, Puglisi EV, Marshall RA, Aitken CE, Korlach J, Ehrenberg M, Puglisi JD. Cell Rep. 2013 Feb 21;3(2):497-508. Pubmed
2012
- Unraveling the dynamics of ribosome translocation. Chen J, Tsai A, O’Leary SE, Petrov A, Puglisi JD. Curr Opin Struct Biol. 2012 Dec;22(6):804-14. Pubmed
- Single-molecule analysis of translational dynamics. Petrov A, Chen J, O’Leary S, Tsai A, Puglisi JD. Cold Spring Harb Perspect Biol. 2012 Sep 1;4(9):a011551. Pubmed
- Isolation of NEDDylated proteins in human cells. Leidecker O, Xirodimas DP. Methods Mol Biol. 832:133-40. Pubmed
- Heterogeneous pathways and timing of factor departure during translation initiation. Tsai A, Petrov A, Marshall RA, Korlach J, Uemura S, Puglisi JD. Nature. 2012 Jul 19;487(7407):390-3. Pubmed
- Chen J, Tsai A, Petrov A, Puglisi JD. Nonfluorescent quenchers to correlate single-molecule conformational and compositional dynamics. J Am Chem Soc. 2012 Apr 4;134(13):5734-7. Pubmed
2011
- Petrov A, Kornberg G, O’Leary S, Tsai A, Uemura S, Puglisi JD. Dynamics of the translational machinery. Curr Opin Struct Biol. 2011 Feb;21(1):137-45. Pubmed
Organisation nucléaire de la transcription dans les embryons
 Albert TSAI
Albert TSAI
Chef d’équipe (CRCN CNRS)
aa
Membre de l’équipe
(Chercheur CRCN) +33 (0)4 34 35 95 05 |
|
(IE-Recherche) +33 (0)4 34 35 95 01 |
|
(Doctorant) +33 (0)4 34 35 95 01 |
|
(Chercheur) +33 (0)4 34 35 95 01 |
|
(Stagiaire) +33 (0)4 34 35 95 01 |
aa
 aa
aa
Contactez notre équipe
Replace the name and address below with that of the member to contact
Prénom.nom@crbm.cnrs.fr
aa
VIDEO
aa